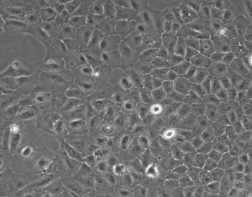
人前列腺癌细胞DU 145
BLUEFBIO™ Product Sheet
|
细胞名称 |
人前列腺癌细胞DU145 |
|
|
|
货物编码 |
BFN60700106 |
||
|
产品规格 |
T25培养瓶x1 |
1.5ml冻存管x2 |
|
|
细胞数量 |
1x10^6 |
1x10^6 |
|
|
保存温度 |
37℃ |
-198℃ |
|
|
运输方式 |
常温保温运输 |
干冰运输 |
|
|
安全等级 |
1 |
||
|
用途限制 |
仅供科研用途 1类 |
||
验收细胞注意事项
1、收到人前列腺癌细胞DU145细胞,请查看瓶子是否有破裂,培养基是否漏出,是否浑浊,如有请尽快联系。
2、收到人前列腺癌细胞DU145细胞,如包装完好,请在显微镜下观察细胞。,由于运输过程中的问题,细胞培养瓶中的贴壁细胞有可能从瓶壁中脱落下来,显微镜下观察会出现细胞悬浮的情况,出现此状态时,请不要打开细胞培养瓶,应立即将培养瓶置于细胞培养箱里静止 3-5 小时左右,让细胞先稳定下,再于显微镜下观察,此时多数细胞会重新贴附于瓶壁。如细胞仍不能贴壁,请用台盼蓝染色法鉴定细胞活力,如台盼蓝染色证实细胞活力正常请按悬浮细胞的方法处理。
3、收到人前列腺癌细胞DU145细胞后,请镜下观察细胞,用恰当方式处理细胞。若悬浮的细胞较多,请离心收集细胞,接种到一个新的培养瓶中。弃掉原液,使用新鲜配制的培养基,使用进口胎牛血清。刚接到细胞,若细胞不多时 血清浓度可以加到 15%去培养。若细胞迏到 80%左右 ,血清浓度还是在 10%。
4、收到人前列腺癌细胞DU 145细胞时如无异常情况 ,请在显微镜下观察细胞密度,如为贴壁细胞,未超过80%汇合度时,将培养瓶中培养基吸出,留下 5-10ML 培养基继续培养:超过 80%汇合度时,请按细胞培养条件传代培养。如为悬浮细胞,吸出培养液,1000 转/分钟离心 3 分钟,吸出上清,管底细胞用新鲜培养基悬浮细胞后移回培养瓶。
5、将培养瓶置于 37℃培养箱中培养,盖子微微拧松。吸出的培养基可以保存在灭菌过的瓶子里,存放于 4℃冰箱,以备不时之需。
6、24 小时后,人前列腺癌细胞DU 145细胞形态已恢复并贴满瓶壁,即可传代。(贴壁细胞)将培养瓶里的培养基倒去,加 3-5ml(以能覆盖细胞生长面为准)PBS 或 Hanks’液洗涤后弃去。加 0.5-1ml 0.25%含 EDTA 的胰酶消化,消化时间以具体细胞为准,一般 1-3 分钟,不超过 5 分钟。可以放入37℃培养箱消化。轻轻晃动瓶壁,见细胞脱落下来,加入 3-5ml 培养基终止消化。用移液管轻轻吹打瓶壁上的细胞,使之完全脱落,然后将溶液吸入离心管内离心,1000rpm/5min。弃上清,视细胞数量决定分瓶数,一般一传二,如细胞量多可一传三,有些细胞不易传得过稀,有些生长较快的细胞则可以多传几瓶,以具体细胞和经验为准。(悬浮细胞)用移液管轻轻吹打瓶壁,直接将溶液吸入离心管离心即可。
7、贴壁细胞 ,悬浮细胞。严格无菌操作。换液时,换新的细胞培养瓶和换新鲜的培养液,37℃,5%CO2 培养。
特别提醒: 原瓶中培养基不宜继续使用,请更换新鲜培养基培养。